# SARS-CoV-2, COVID-19, saliva, kit de diagnóstico
resumen
rápido y preciso SARS-CoV-2 pruebas de diagnóstico es fundamental para controlar el COVID-19 pandemia. El estándar de oro actual para COVID-19 el diagnóstico es en tiempo real RT-PCR detección de SARS-CoV-2 de hisopos nasofaríngeos. Sin embargo, la baja sensibilidad, los riesgos de exposición para los trabajadores de la salud y la escasez mundial de hisopos y equipo de protección personal requieren la validación de nuevos enfoques de diagnóstico. la saliva es un candidato prometedor para SARS-CoV-2 diagnóstico porque (1) La recolección es mínimamente invasiva y se puede autoadministrar de manera confiable y (2) la saliva ha mostrado una sensibilidad comparable a los hisopos nasofaríngeos en la detección de otros patógenos respiratorios, incluidos los coronavirus humanos endémicos, en estudios anteriores. Para validar el uso de saliva para SARS-CoV-2 detección, analizamos muestras nasofaríngeas y de saliva confirmado COVID-19 pacientes y muestras autocogidas de trabajadores de la salud en COVID-19 salas. Cuando comparamos SARS-CoV-2 detección de adaptado al paciente nasofaríngeo
y muestras de saliva, encontramos que la saliva produjo una mayor sensibilidad y consistencia de detección a lo largo del curso de la infección. Además, informamos menos variabilidad en automuestra recogida de saliva. en conjunto, nuestros hallazgos demuestran que la saliva es una alternativa viable y más sensible a los hisopos nasofaríngeos y podría habilitar en casa autoadministrado recolección de muestras para precisión a gran escala SARS-CoV-2 pruebas.
Introducción
esfuerzos para controlar el SARS-CoV-2, el nuevo coronavirus causando COVID-19 pandemia, dependen de pruebas diagnósticas rápidas y precisas. Estos las pruebas deben ser ( 1 ) sensible a infecciones leves y asintomáticas para promover un autoaislamiento eficaz y reducir la transmisión en grupos de alto riesgo1 ; ( 2 ) consistente para monitorear confiablemente la progresión de la enfermedad y ayudar a las decisiones clínicas2 ; y ( 3 ) escalable para informar las políticas de salud pública locales y nacionales, como when Las medidas de distanciamiento social se pueden relajar de forma segura. Sin embargo, el SARS-CoV-2 las estrategias de prueba a menudo no cumplen con estos criterios, en parte porque de su dependencia de los hisopos nasofaríngeos como el tipo de muestra ampliamente recomendado para RT-PCR en tiempo real.
Aunque Los hisopos nasofaríngeos se utilizan comúnmente en el diagnóstico de virus respiratorios, ellos muestran una sensibilidad relativamente baja para SARS-CoV-2 detección en la infección temprana y son 2–6 inconsistente durante pruebas en serie . Además, la recolección de hisopos nasofaríngeos causa incomodidad a los pacientes debido a la procedimiento invasividad, lo que limita el cumplimiento de las pruebas repetidas y presenta un riesgo considerable para los trabajadores de la salud, porque puede inducir a los pacientes a estornudar o toser, expulsando virus partículas7 . El procedimiento tampoco es propicio para pruebas a gran escala, porque hay una escasez generalizada de hisopos y equipos de protección personal para el cuidado de la salud trabajadores8 y autocolección de hisopos nasofaríngeos es difícil y menos sensible para la detección de virus 9 . Estos Los desafíos se agravarán aún más a medida que COVID-19 la pandemia se intensifica en los países de bajos ingresos. dadas las limitaciones, un sistema más fiable y menos intensivo en recursos método de recolección de muestras, idealmente uno que se adapte a la auto-recolección en el hogar, se necesita con urgencia. el muestreo de saliva es una alternativa atractiva al hisopado nasofaríngeo, desde recolectar saliva no es invasivo y fácil de autoadministrar. un análisis de concordancia nasofaríngea y saliva para RT-PCR detección de patógenos respiratorios, incluidos dos estacionales
coronavirus humanos, sugiere una sensibilidad de diagnóstico comparable entre las dos muestras 10,11de COVID-19 pacientes y ( 2 ) las muestras de saliva recolectadas por ellos mismos son comparables. los hallazgos preliminares indican que ( 1 ) SARS-CoV-2 se puede detectar desde la saliva 12 tiposmSARS-CoV-2 Sensibilidad de detección a hisopos nasofaríngeos recogidos por el personal sanitario 13trabajadores de leve y subclínico COVID-19 casos . Sin embargo, es fundamental que no exista una evaluación rigurosa de la sensibilidad de SARS-CoV-2 se ha realizado la detección en saliva con respecto a hisopos nasofaríngeos de pacientes hospitalizados durante el curso de COVID-19 infección.
en este estudio, evaluamos SARS-CoV-2 detección en muestras pareadas de hisopos nasofaríngeos y saliva recogidas de COVID-19 pacientes hospitalizados y trabajadores sanitarios asintomáticos en moderado a alto riesgo de COVID-19 exposición. Nuestro Los resultados indican que el uso de saliva para SARS-CoV-2 la detección es más sensible y consistente que usando hisopos nasofaríngeos. En general, demostramos que la saliva debería ser considerado como un tipo de muestra confiable para aliviar COVID-19 pruebas demandas.
resultados
más alto SARS-CoV-2 títulos detectados de saliva que hisopos nasofaríngeos pacientes hospitalizados
Para determinar si la saliva funciona tan bien como los ee.uu. recomendación de los cdc de usar hisopos nasofaríngeos para SARS-CoV-2 diagnósticos, recolectamos muestras clínicas de 44 COVID-19 participantes del estudio de pacientes hospitalizados ( tabla 1 ). Este cohorte representa un rango de COVID-19 pacientes con enfermedad grave, con 19 (43%) que requieren cuidados intensivos, 10 (23%) que requieren ventilación mecánica y 2 (5%) fallecido el 5 de abril 2020. utilizando los EE. UU. cdc SARS-CoV-2 RT-PCR ensayo, probamos 121 saliva recolectada por uno mismo o para la atención médica administrada por el trabajador hisopos nasofaríngeos esta cohorte. Nosotros encontró una fuerte concordancia entre los EE. UU. cdc “N1” y “N2” cebador-sonda conjuntos ( datos extendidos Fig. 1) y, por tanto, los títulos de virus calculados (virus copias / mL) utilizando sólo el “N1” conjunto. Desde todas las muestras positivas analizadas ( norte = 46 nasofaríngeas, 37 saliva), encontramos que la media geométrica de los títulos de virus de saliva eran aproximadamente 5⨉ mayor que hisopos nasofaríngeos ( pags < 0,05, Mann-Whitney prueba; Fig. 1a ). Cuando limitando nuestro análisis a sólo emparejado por paciente muestras nasofaríngeas y de saliva ( norte = 38 para cada tipo de muestra), encontramos que SARS-CoV-2 títulos de saliva fueron significativamente más altos que hisopos nasofaríngeos ( pags = 0,0001, Wilcoxon prueba; Fig. 1b ). Además, detectamos SARS-CoV-2 de la saliva pero no los hisopos nasofaríngeos ocho muestras coincidentes (21%), mientras que solo detectamos SARS-CoV-2 de hisopos nasofaríngeos y no saliva tres muestras emparejadas (8%; Fig. 1c ). En general, encontramos mayor SARS-CoV-2 títulos de saliva que hisopos nasofaríngeos hospital pacientes hospitalizados
cuadro 1. COVID-19 características de la cohorte de pacientes hospitalizados
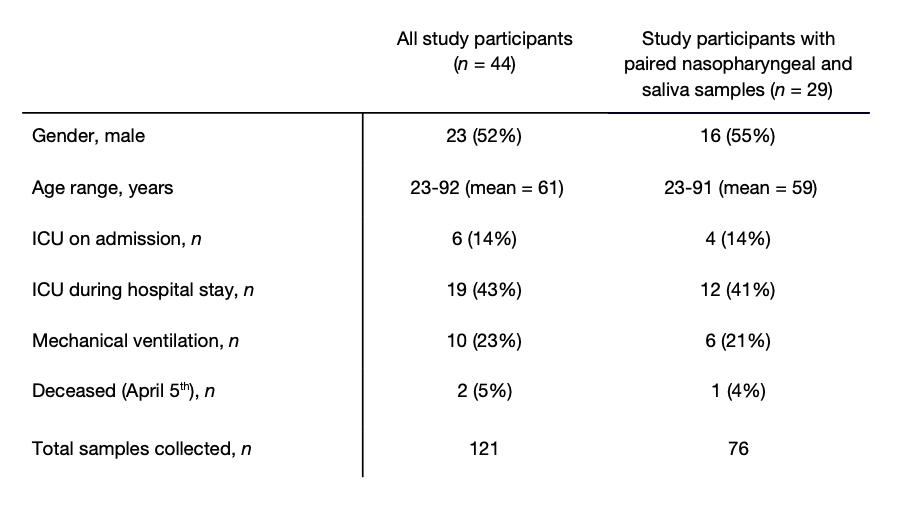
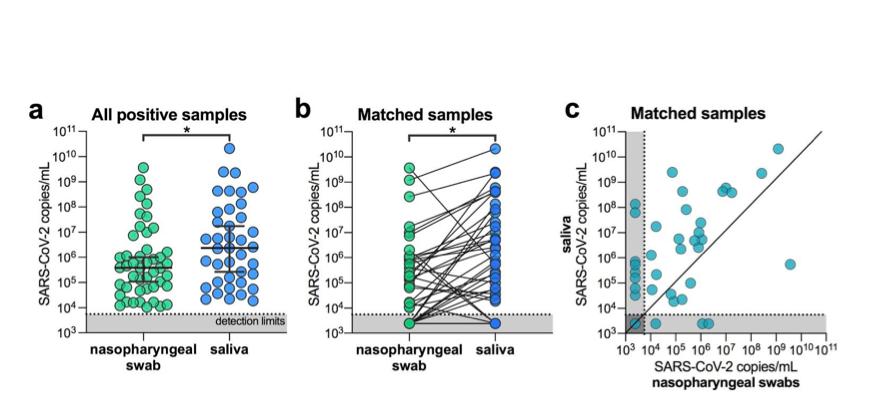
figura 1. SARS-CoV-2 los títulos son más altos en la saliva que hisopos nasofaríngeos hospital pacientes hospitalizados ( un ) todos los hisopos nasofaríngeos positivos ( norte = 46) y muestras de saliva ( norte = 39) fueron comparados por un Mann-Whitney prueba ( pags < 0,05). las barras representan la mediana y 95% CI. Nuestro límites de detección del ensayo para SARS-CoV-2 usando el cdc us “N1” el ensayo se encuentra en el umbral de ciclo 38, que corresponde a 5610 virus copias / mL de la muestra (mostrada como línea de puntos y área gris ). ( segundo ) muestras emparejadas de pacientes ( norte = 38), representados por las líneas de conexión, se compararon mediante un Wilcoxon prueba de prueba ( pags < 0,05). ( C ) muestras emparejadas de pacientes ( norte = 38) también se representan en un gráfico de dispersión. Todos los datos utilizados para generar esta figura, incluidos los umbrales de ciclo sin procesar, se pueden encontrar en datos suplementarios 1 . datos extendidos Fig. 1muestra la correlación entre el ensayo CDC de EE. UU. “N1” y “N2” resultados.
menos temporal SARS-CoV-2 variabilidad cuando prueba de saliva pacientes hospitalizados
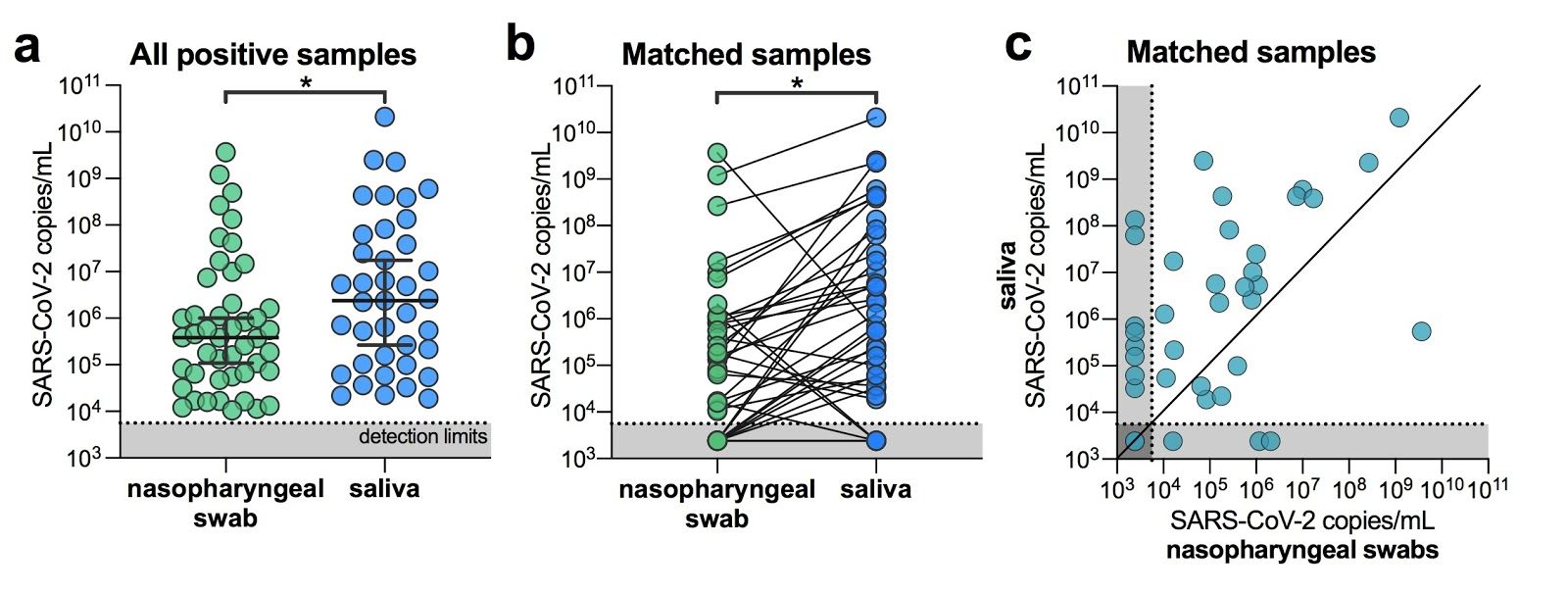
como temporal SARS-CoV-2 pruebas de diagnóstico de Se informa que los hisopos nasofaríngeos son 2,3variable, probamos muestras longitudinales nasofaríngeas y de saliva pacientes hospitalizados para determinar qué tipo de muestra proporcionó resultados más consistentes. Desde 22 participantes con múltiples hisopos nasofaríngeos y 12 participantes con múltiples muestras de saliva, encontramos que SARS-CoV-2 los títulos generalmente disminuyeron en ambos tipos de muestras después de la fecha informada de inicio de los síntomas ( Fig. 2a ). Nuestro Los resultados de los hisopos nasofaríngeos son consistentes con 2,3informes anteriores de la variable SARS-CoV-2 títulos y resultados : encontramos 5 instancias donde un participante El hisopado nasofaríngeo fue negativo para SARS-CoV-2 seguido de un resultado positivo durante la siguiente colección (5 / 33 repeticiones, 33%; Fig. 2b ) . en colecciones longitudinales de saliva 12 pacientes, sin embargo, no hubo casos en los que una muestra diera negativo y luego fuera seguida de un resultado positivo. Dado que los resultados negativos verdaderos de las pruebas son importantes para que los médicos realicen un seguimiento de las mejoras de los pacientes y para tomar decisiones sobre las altas, nuestros datos sugieren que la saliva es un tipo de muestra más consistente hisopos nasofaríngeos para monitorear cambios temporales en SARS-CoV-2 títulos.
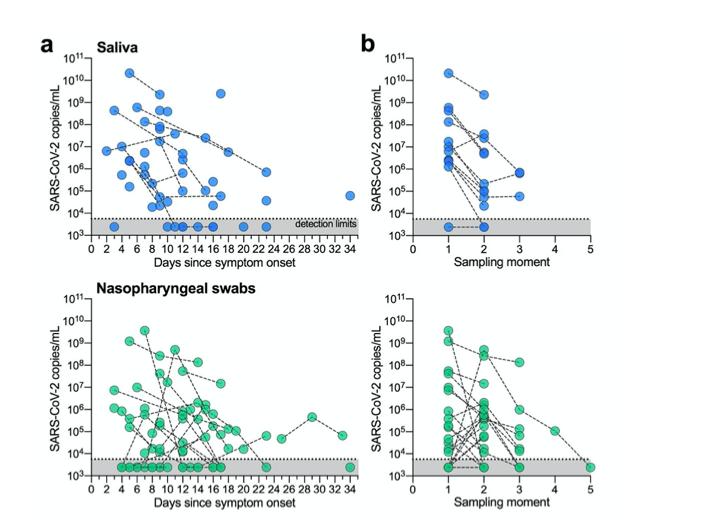
figura 2: SARS-CoV-2 la detección es menos variable entre las recolecciones de muestras repetidas con saliva. (a) longitudinal SARS-CoV-2 títulos de saliva o hisopos nasofaríngeos se muestran como días desde síntoma inicio. cada círculo representa una muestra separada, que está conectada a muestras adicionales de el mismo paciente por una línea discontinua Nuestro límites de detección del ensayo para SARS-CoV-2 usando el cdc us “N1” el ensayo se encuentra en el umbral de ciclo 38, que corresponde a 5610 virus copias / mL de la muestra (mostrada como línea de puntos y área gris ). ( segundo ) Los datos también se muestran por momento de muestreo (secuencial recopilación tiempo) para resaltar las diferencias en los títulos de virus entre los puntos de recolección. Todos los datos utilizados para generar esta figura, incluidos los umbrales de ciclo sin procesar, se pueden encontrar en datos suplementarios 1 .
más consistente auto-muestreo de trabajadores de la salud que usan saliva
validación de saliva para la detección de subclínicos SARS-CoV-2 infecciones podría probar transformador tanto para el diagnóstico remoto de pacientes como para la vigilancia del personal sanitario. Para investigamos esto, inscribimos a 98 trabajadores sanitarios asintomáticos nuestro estudio y saliva recolectada y / o hisopos nasofaríngeos en promedio cada 2.9 días (rango = 1-8 días, Tabla 2 ). Para fecha, hemos detectado SARS-CoV-2 en saliva de dos trabajadores de la salud que resultaron negativos por hisopos nasofaríngeos utilizando tanto el CDC de EE. UU. “N1” y “N2” pruebas y no informó ningún síntoma. La saliva de uno de estos de nuevo dieron positivo junto con un hisopo nasofaríngeo negativo coincidente repita la prueba 2 días después. títulos de virus asistencia sanitaria asintomática trabajadores " la saliva es menor que que normalmente detectamos de pacientes hospitalizados sintomáticos ( Fig. 3a ), lo que probablemente respalda la falta de síntomas.
Nuestro datos limitados apoyan que la saliva puede ser más sensible para detectar asintomáticos o presintomáticos infecciones; sin embargo, se necesita un tamaño de muestra mayor para confirmar. ya que la inconsistencia de la muestra de hisopos nasofaríngeos puede ser uno de los problemas potenciales de falsos negativos ( Fig. 2), el seguimiento de un control interno para la recogida adecuada de muestras, la rnasa P humana, puede proporcionar una técnica de evaluación alternativa. mientras que la detección de la rnasa p humana fue mejor saliva en las cohortes de pacientes hospitalizados y trabajadores de la salud ( Fig. 3b ) , esto por sí solo puede no indicar una mejor detección de virus. lo que es más importante, encontramos que la detección de la rnasa p humana era más variable de hisopos nasofaríngeos recogidos de pacientes hospitalizados ( pags = 0,0001, prueba f para varianzas) y auto-recolectados de trabajadores de la salud ( pags = 0,0002; Fig. 3b ). Nuestro Los resultados sugieren que la saliva también puede ser una alternativa apropiada, y quizás más sensible, a los hisopos nasofaríngeos para la detección de síntomas asintomáticos o pre-sintomáticos SARS-CoV-2 infecciones.
cuadro 2. cohorte de trabajadores sanitarios
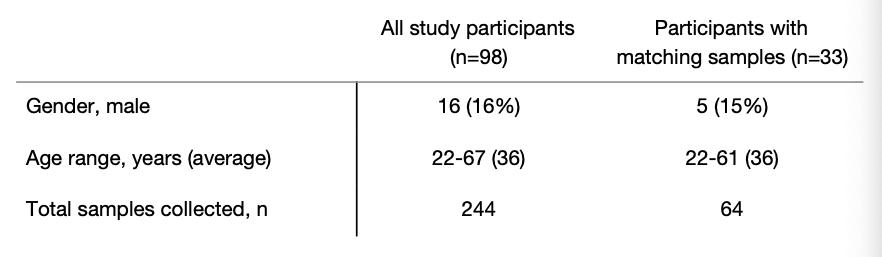
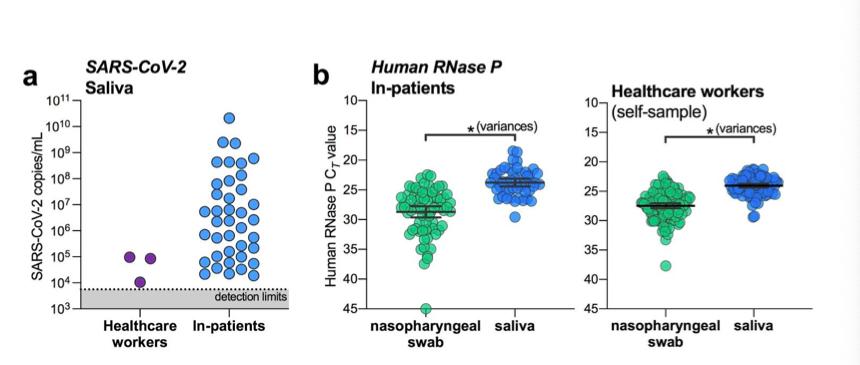
figura 3. la saliva es una alternativa para SARS-CoV-2 proyección de trabajadores sanitarios y casos asintomáticos. ( un ) SARS-CoV-2 títulos medidos de la saliva de los trabajadores sanitarios y de los pacientes hospitalizados. Nuestro límites de detección del ensayo para SARS-CoV-2 usando el cdc us “N1” el ensayo se encuentra en el umbral de ciclo 38, que corresponde a 5610 virus copias / mL de la muestra (mostrada como línea de puntos y área gris ). ( segundo ) RT-PCR umbrales de ciclo (Ct) valores de rnasa P humana y control interno para la recogida de muestras, pacientes hospitalizados (izquierda panel) o trabajadores de la salud (derecha panel) se compararon por varianzas utilizando la prueba f ( pags = 0,0001 para pacientes hospitalizados; pags = 0,0002 para trabajadores sanitarios). Todos los datos utilizados para generar esta figura, incluidos los umbrales de ciclo sin procesar, se pueden encontrar en datos suplementarios 1 .
discusión
Nuestro estudio demuestra que la saliva es una alternativa viable y preferible a los hisopos nasofaríngeos para SARS-CoV-2 detección. Nosotros encontró que la sensibilidad de SARS-CoV-2 detección de la saliva es comparable, si no superior, a los hisopos nasofaríngeos en la hospitalización temprana y es más consistente durante hospitalización prolongada y recuperación. Además, la detección de SARS-CoV-2 de la saliva de dos trabajadores sanitarios asintomáticos a pesar de que los hisopos nasofaríngeos coincidentes negativos indican que la saliva también puede ser una alternativa viable para identificar infecciones leves o subclínicas. Con mayor validación, implementación generalizada del muestreo de saliva ser transformador para la salud pública esfuerzos: saliva autocolección niega la necesidad de asistencia sanitaria directa trabajador-paciente interacción, una fuente de 14–16 varios cuellos de botella importantes en las pruebas y el riesgo general de infección nosocomial, y alivia la demanda de suministro de hisopos y equipos de protección personal.
Las cargas de virus AsSARS-CoV-2 difieren entre casos leves y graves , una limitación de nuestro estudio es el enfoque principal en COVID-19 pacientes hospitalizados, muchos con enfermedades graves. mientras que se requieren más datos para comparar de manera más rigurosa la eficacia de la saliva en el entorno hospitalario con una etapa más temprana de la infección, los hallazgos dos estudios recientes apoyan su potencial para 13,18 detectando SARS-CoV-2 de tanto individuos asintomáticos como ambulatorios . como 12 se ha detectado un virus infeccioso de la saliva de COVID-19 pacientes, verificando la relación entre las copias del genoma del virus y las partículas de virus infecciosas en la saliva de 19 presintomático Los individuos jugarán un papel clave en la comprensión de la dinámica de 1,20 transmisión asintomática.
derivado de los prometedores resultados para SARS-CoV-2 detección en asintomáticos 13
individuos, el ensayo de detección asalivaSARS-CoV-2 ya ha obtenido la aprobación a través del
nos. autorización de uso de emergencia de la administración de alimentos y medicamentos . Para satisfacen las crecientes demandas de pruebas, sin embargo, nuestros hallazgos respaldan la necesidad de una validación e implementación inmediata de la saliva para SARS-CoV-2 diagnósticos en laboratorios clínicos certificados.
métodos
ética
todos los participantes del estudio fueron inscritos y seleccionados de acuerdo con la universidad de yale aprobada por HIC protocolo # 2000027690. Los datos demográficos, los datos clínicos y las muestras solo se recopilaron después de que el participante del estudio reconoció que ellos había entendido el protocolo del estudio y firmado el consentimiento informado. toda la información y las muestras de los participantes se recopilaron en asociación con los identificadores del estudio.
inscripción de participantes
pacientes hospitalizados
pacientes ingresados en el hospital de yale new haven (un centro médico de atención terciaria de 1541 camas en new Haven, CT, EE. UU.), que dieron positivo para SARS-CoV-2 por nasofaringe y / o hisopo orofaríngeo (CDC aprobado ensayo) fueron invitados a inscribirse en el estudio de investigación. Los criterios de exclusión fueron menores de 18 años, no inglés evidencia oral y clínica, radiológica o de laboratorio de un no infeccioso causa de fiebre o síntomas respiratorios o un confirmado microbiológicamente fuente infecciosa (por ejemplo gastrointestinal, urinaria, cardiovascular) que no sea tracto respiratorio para síntomas y sin sospecha de COVID-19 infección.
trabajadores de la salud
trabajadores sanitarios asintomáticos (p. ej., sin fiebre ni síntomas respiratorios) con exposición ocupacional a pacientes con COVID-19 fueron invitados a inscribirse en el estudio. La participación en el estudio permitió una vigilancia activa para garantizar la detección temprana después de la exposición y proteger aún más a otros trabajadores sanitarios y pacientes.
coleccion de muestra
pacientes hospitalizados
Se obtuvieron muestras nasofaríngeas y de saliva cada tres días a lo largo de curso clínico. enfermeras tituladas tomaron muestras nasofaríngeas utilizando el transporte viral universal bd (UVT) sistema. El flexible, mini-tip se pasó el hisopo a través del paciente fosa nasal hasta se alcanzó la nasofaringe posterior, se dejó en su lugar durante varios segundos para absorber las secreciones y luego se extrajo lentamente mientras se giraba. El hisopo se colocó en el medio de transporte viral estéril (total volumen 3 ml) y sellado de forma segura. Las muestras de saliva fueron recogidas por el paciente. Sobre al despertar, se pidió a los pacientes que evitaran la comida, el agua y el cepillado de los dientes hasta se recogió la muestra. se pidió a los pacientes que escupieran repetidamente en una taza de orina estéril hasta aproximadamente un tercio lleno de líquido (excluidas las burbujas), antes de cerrarlo de forma segura. todas las muestras se almacenaron a temperatura ambiente y se transportaron al laboratorio de investigación de la escuela de salud pública de yale dentro de las 5 horas posteriores a la recolección de la muestra.
trabajadores de la salud
se pidió a los trabajadores de la salud que recogieran un hisopado nasofaríngeo y una muestra de saliva cada tres días durante un período de 2 semanas. las muestras se almacenaron a + 4 ° C hasta siendo transportado al laboratorio de investigación.
SARS-CoV-2 detección
a la llegada al laboratorio de investigación, total nucleico se extrajo ácido 300 μl de medios de transporte viral el hisopo nasofaríngeo o 300 μl de saliva entera usando el MagMAX Viral / Patógeno Nucleico kit de aislamiento de ácido (ThermoFisher Scientific) siguiendo el fabricante protocolo y eluido en 75 μl de elución tampón. Para SARS-CoV-2 ARN 21,22 detección, 5 μl de la plantilla de ARN se probó como se describió anteriormente, utilizando el CDC de EE. UU. en tiempo real RT-PCR cebador / sonda conjuntos para 2019-nCoV_N1 y 2019-nCoV_N2 y la rnasa humana p (RP) como control de extracción. las muestras se clasificaron como positivas para SARS-CoV-2 cuando ambos N1 y N2 cebador-sonda se detectaron conjuntos <38 c . copias de virus fueron t cuantificado utilizando un 10 veces curva estándar de dilución de las transcripciones de ARN que previamente 21generado . como resultados de N1 y N2 eran comparables ( datos extendidos Fig. 1), todas las copias de virus se muestran calculadas utilizando el N1 cebador-sonda conjunto.
análisis estadístico
Los análisis estadísticos se realizaron en GraphPad prisma 8.0.0 como se describe en Resultados.
expresiones de gratitud
Nosotros agradecer a los participantes del estudio por su tiempo y compromiso con el estudio. Nosotros gracias a todos los miembros del equipo clínico de Yale-New hospital refugio para su dedicación y trabajo que hicieron posible este estudio. Nosotros también agradezco S. taylor y P. jack para discusiones técnicas.
fondos
El estudio fue financiado en parte por el instituto yale para la salud global. Los autores correspondientes tuvieron acceso completo a todos los datos del estudio y tuvieron la responsabilidad final de la decisión de enviar para su publicación.
datos extendidos
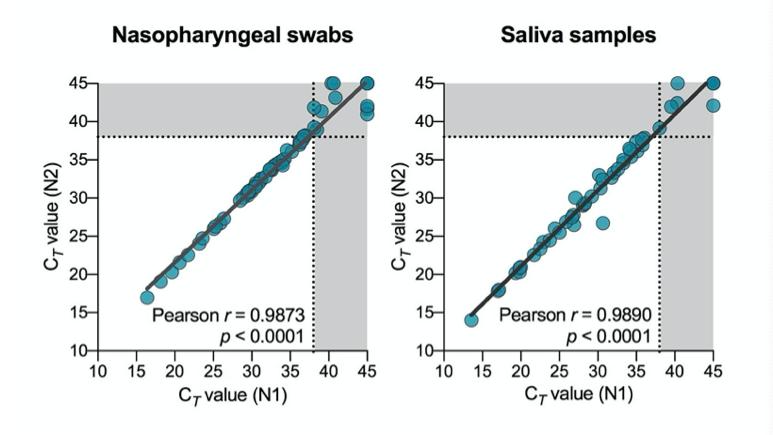
datos extendidos Fig. 1. concordancia entre SARS-CoV-2 detección usando us cdc “N1” y “N2” conjuntos de cebadores y sondas.ct = RT-PCR ciclo umbral. la línea de puntos y las áreas grises indican los límites de detección.
referencias
1. Kimball, A. et al. asintomático y presintomático SARS-CoV-2 infecciones en residentes de un centro de enfermería especializada de cuidados a largo plazo - King County, Washington, marzo 2020. MMWR Morb. Mortal. Wkly. Rep. 69, 377–381 (2020).
2. Wölfel, R. et al. Evaluación virológica de pacientes hospitalizados con COVID-2019. naturaleza (2020) doi: 10.1038 / s41586-020-2196-x.
3. Zou, L. et al. SARS-CoV-2 carga viral en muestras respiratorias superiores de pacientes infectados. N. Engl. J. Med. 382 , 1177–1179 (2020).
4. Zhao, J. et al. respuestas de anticuerpos a SARS-CoV-2 en pacientes de nuevo coronavirus enfermedad 2019. Clin. Infectar. Dis. (2020) doi: 10.1093 / cid / ciaa344.
5. Xie, X. et al. tórax para típico 2019-nCoV Neumonía: relación con negativo RT-PCR Pruebas. radiología 200343 (2020).
6. Wang, W. et al. detección de SARS-CoV-2 en diferentes tipos de muestras clínicas. JAMA (2020) doi: 10.1001 / jama.2020.3786.
7. Para, K. K.-W. et al. perfiles temporales de carga viral en muestras de saliva orofaríngea posterior y respuestas de anticuerpos séricos durante Infección por SARS-CoV-2: un estudio de cohorte observacional.lanceta Infectar. Dis. (2020) doi: 10.1016 / S1473-3099 (20) 30196-1.
8. CDC. recomendaciones provisionales de prevención y control de infecciones para pacientes con sospecha o confirmación de coronavirus enfermedad 2019 (COVID-19) en el ámbito de la salud Centros de Control y Prevención de Enfermedades https: / / www.cdc.gov / coronavirus / 2019-ncov / hcp / recomendaciones-de-control-de-infecciones.h tml? CDC_AA_refVal = https% 3A% 2F% 2Fwww.cdc.gov% 2Fcoronavirus% 2F2019-ncov % 2Finfection-control% 2Fcontrol-recommended.html (2020).
9. Dhiman, N. et al. eficacia de recogida por el paciente hisopos para pruebas de influenza.mayo Clin. Proc. 87, 548–554 (2012).
10. Kim, Y.-G. et al. comparación entre muestras de hisopos de saliva y nasofaríngeas para la detección de virus respiratorios por multiplex inversa Transcripción-PCR. J. Clin. Microbiol. 55, 226–233 (2017).
11. Wyllie, A. L. et al. vigilancia molecular del transporte nasofaríngeo de estreptococos pneumoniae en niños vacunados con vacunas neumocócicas de polisacáridos conjugados. Ciencia. Rep. 6, 23809 (2016).
12. Para, K. K.-W. et al. detección constante de 2019 novela Coronavirus en Saliva. Clin. Infectar. Dis. (2020) doi: 10.1093 / cid / ciaa149.
13. Kojima, N. et al. El líquido oral y los hisopos nasales recolectados por el médico demuestran una sensibilidad comparable a los hisopos nasofaríngeos recolectados por el médico para Covid-19 Detección. medRxiv 2020.04.11.20062372 (2020).
14. Tran, K., Cimón, K., Severn, M., Pessoa-Silva, C. L. & Conly, J. procedimientos generadores de aerosoles y riesgo de transmisión de infecciones respiratorias agudas a los trabajadores sanitarios: una revisión sistemática. Más uno 7, e35797 (2012).
15. Judson, S. D. & Munster, V. J. transmisión nosocomial de virus emergentes vía Generador de aerosoles médicos Procedimientos. virus 11, (2019).
16. Wang, D. et al. características clínicas de 138 pacientes hospitalizados Con 2019 novela Infectados por coronavirus neumonía en Wuhan, China. JAMA (2020) doi: 10.1001 / jama.2020.1585.
17. Liu, Y. et al. dinámica viral en casos leves y graves de COVID-19. lanceta Infectar. Dis. (2020) doi: 10.1016 / S1473-3099 (20) 30232-2.
18. nos. comida & fármaco Administración. autorización de uso de emergencia acelerada (EUA) resumen SARS-CoV-2 ensayo (Rutgers genómica clínica Laboratorio). https: / / www.fda.gov / media / 136875 / descargar.
19. Lauer, S. A. et al. El período de incubación de Coronavirus enfermedad 2019 (COVID-19) Desde denunciado públicamente confirmado Casos: estimación y Aplicación. Ann. Pasante Med.
Fuente: https: / / www.medrxiv.org / contenido / 10.1101 / 2020.04.16.20067835v1.full.pdf
AnneL.Wyllie1 *, JohnFournier2, ArnauCasanovas-Massana1, MelissaCampbell2, MariaTokuyama3, Pavithra Vijayakumar4 , Bertie Geng4 , M. catherine Muenker1 , Adán J. Moore1 , Chantal B.F. Vogels1 , María E. Petrone1 , Isabel M. Ott5, Peiwen Lu3 , Arvind Venkataraman3 , Alice Lu-Culligan3 , jonathan Klein3 , Rebecca Earnest1 , Michael Simonov6 , Rupak Datta2 , Ryan Handoko2 , Nida Naushad2 , Lorenzo R. Sewanan2 , Jordania Valdez2 , elizabeth B. Blanco1 , sarah Lapidus1 , Chaney C. Kalinich1 , Xiaodong Jiang3 , daniel J. Kim3 , Eriko Kudo3 , melissa Linehan3 , Tianyang Mao3 , Miyu Moriyama3 , ji Eun Oh3 , Annsea Park3 , Julio Silva3 , Eric Song3 , Takehiro Takahashi3 , Manabu Taura3 , Orr-El Weizman3 , patrick Wong3 , Yexin Yang3 , santos Bermejo7 , Camila Odio8 , Saad B. Omer1,2,9,10, charles S. Dela Cruz7 , ShelliFarhadian2, RichardA.Martinello2,7,11, AkikoIwasaki3,12, NathanD.Grubaugh1 # *, AlbertI.Ko1 # *
1 departamento de epidemiología de enfermedades microbianas, escuela de salud pública de yale, new Haven, ct 06510, EE.UU. 2 departamento de medicina, sección de enfermedades infecciosas, escuela de medicina de yale, new Haven, CT, 06510, EE. UU.
3 Departamento de Inmunobiología, Escuela de Medicina de Yale, New Haven, CT, 06510, EE. UU.
4 departamento de obstetricia, ginecología y ciencias reproductivas, escuela de medicina de yale, new Haven, CT, 06510, EE. UU.
5 departamento de ecología y biología evolutiva, Universidad de yale, New Haven, ct 06520, EE. UU.
6 programa de investigación traslacional aplicada, yale School of Medicine, new Haven, CT, 06510, EE. UU.
7 Departamento de Medicina Interna, Sección de Medicina Pulmonar, Cuidados Críticos y del Sueño, Facultad de Medicina de Yale, New Haven, CT, 06510, EE. UU.
8 Departamento de Medicina, Grupo médico del noreste, Yale-New haven Health, New Haven, CT 06510, EE. UU.
9 instituto yale de salud global, new Haven, ct 06510, EE. UU.
10 yale school of Nursing, new Haven, ct 06510, EE. UU.
11 Departamento de Prevención de Infecciones, Yale-New haven Health, new Haven, ct 06520
12 Instituto Médico Howard Hughes, New Haven, CT 06510, EE. UU.
# autores senior conjuntos
* Correspondencia: anne.wyllie@yale.edu (ALW); nathan.grubaugh@yale.edu (NDG); albert.ko@yale.edu (AIK)